COFECUB-CAPES project with Brazil (2023-2026)
A new COFECUB-CAPES project has started in 2023 for three years between the Capsid Team, the Catholic University of Brasilia (UCB for « Universidade Católica de Brasília ») and the State University of Maringá (UEM for « Universidade Estadual de Maringá »). This project entitled « New drugs against invasive fungal infections: from hits to optimised leads through machine learning » wants to continue the previous work of Bernard Maigret from the Capsid team with microbiologists at UCB and UEM.
In this context, I have the great opportunity to spend a few days in Brasilia and in Maringá from 19 to 30 November 2023.
I will give two seminars at UCB and one at UEM and also propose several tutorials or master classes for interested students from these two universities.
Here below are the documents required to follow the master classes.
Tutorial GOLD – Flexible docking with GOLD through the Galaxy Interface
Student guide GOLD 29 nov 2023 (pdf, 512K)
Tutorial Pharmit – A powerful interface for pharmacophore-based virtual screening
Student guide PHARMIT 23 nov 2023 (pdf, 465K)
Tutorial WEKA – Introduction to Machine Learning
Slides : a smooth introduction to classification (pdf, 0.9 M)
Tutorial GKBox – Introduction to the exploration of Knowledge Graphs using Neo4J
Enseignement (2021-2022)
CMI : Cursus Master Ingénieur BioSanté: UE6.71, Atelier ICE-Bioinfo
Télécharger Artemis
Fichiers CP002215_fasta.fasta, CP002215_1-genome.txt, CP002215_1-resultatsIceFinder.txt
Projets de recherche en cours
PROJET GraphScore-2 (Contrat d’interface avec le CHRU Nancy-> dec 2020)
L’objectif de ce projet est de noter des sous-graphes extraits d’un réseau d’interactions complexes, en lien avec des recherches cliniques visant à découvrir de nouveaux biomarqueurs de l’insuffisance cardiaque. Des scores de graphes originaux seront définis sur la base des connaissances disponibles sur les interactions qui composent ces graphes et par une estimation statistique de significativité au sein du réseau complexe. Ces scores seront optimisés individuellement ou en combinaison pour leur capacité à discriminer les sous-graphes reliant les pathologies de l’insuffisance cardiaque à des biomarqueurs connus. Ils seront ensuite appliqués aux sous-graphes correspondant à des biomarqueurs candidats afin de les classer par ordre de pertinence.
In July 2019, Amina Ahmed-Nacer has been recruited on a Region-CHRU postdoc offer to work with me on this project.
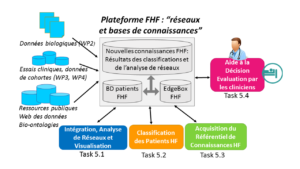
PROJET FIGHT-HF (2015-2020)
FIGHT-HF is for FIGHT Heart Failure, in French « Combattre l’insuffisance cardiaque ». Ce projet de recherche hospitalo universitaire (RHU) a été construit par la Fédération Hospitalo-Universitaire CARTAGE (Cardiac and Arterial Ageing) du CHRU Nancy Brabois. Le Loria (MD Devignes et Malika Smaïl-Tabbone) coordonne le WP5 : « Network-based analysis and integration » dans lequel des recherches sont menées autour d’une plateforme en science des réseaux pour intégrer les connaissances sur l’insuffisance cardiaque ainsi que les données des cohortes étudiées dans le projet, afin de proposer une aide à la décision pour le diagnostic, le traitement et la prévention des différentes formes d’insuffisance cardiaque.
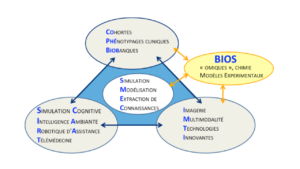
CPER ITM2P (2015-2020)
Le contrat de plan Etat-Région Santé 2015-2020 est piloté par l’INSERM. ITM2P est pour Innovations Technologiques, Modélisation et Médecine Personnalisée. Ce projet est construit autour de 4 plateformes : SMEC, SCIARAT, IMTI et COPHEBIOB (voir schéma ci-dessous), en interaction avec une 5ème plateforme (BIOS) qui ne dépend pas directement du CPER ITM2P. La plateforme SMEC (Simulation, Modélisation et Extraction de Connaissances) est coordonnée par Malika Smaïl-Tabbone et MD Devignes (Loria), Eliane Albuisson (IECL) et François Dehez (SRSMC).
Projets terminés
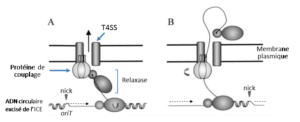
Projet LUE CITRAM (2017-2019)
Ce projet Lorraine Université d’Excellence étudie la Conception d’Inhibiteurs du Transfert de Résistances aux agents Anti-Microbiens. Il est porté par la Pr. Nathalie Leblond-Bourget du laboratoire DynAMic (UMR 1128 INRA Université de Lorraine). La partie Loria est coordonnée par MD Devignes et concerne d’une part le criblage virtuel d’inhibiteurs du site actif de la relaxase du système ICESt3, d’autre part la modélisation des interactions entre cette relaxase et l’ADN qui est transféré d’une cellule à l’autre.